搜索
关闭 X


陈海峰研究团队在组蛋白协同修饰研究中取得新进展
发布时间 :2016-04-22
阅读次数 :2265
近日,Nature系列刊物scientific reports在线发布了hbs红宝石平台陈海峰研究团队的文章“Synergistic Modification Induced Specific Recognition between Histone and TRIM24 via Fluctuation Correlation Network Analysis”,该研究首次采用动态相关网络分析的方法研究了组蛋白协同修饰与TRIM24蛋白的特异性识别。该研究成果是与上海交通大学医学院附属仁济医院以及加州大学尔湾分校合作完成的。
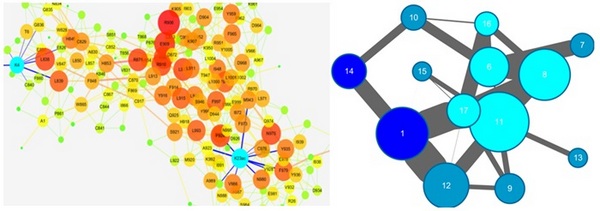
组蛋白可以发生甲基化、乙酰化、磷酸化、泛素化等不同的修饰,从而调控基因的表达;不同的组蛋白修饰能够被对应的组蛋白“reader”识别和结合,然而这些蛋白是如何特异识别特定类型的组蛋白修饰,目前的研究还尚不清楚。
研究利用分子动力学模拟结合动态网络分析的方法,通过构建不同的组蛋白修饰体系,系统的分析了与乳腺癌相关的“reader”蛋白TRIM24与组蛋白修饰的关系。研究发现TRIM24蛋白在结合不同位点和不同类型的组蛋白修饰后构象和蛋白内部网络存在差异显著,在动态网络以及社区网络分类的基础上提出了“协同修饰诱导识别”的理论,对于靶向“reader”蛋白的药物发现具有重要的指导意义。同时,这一方法对于生物大分子间的别构以及信号传导等机制的研究也具有重要意义。


 首页
首页

 学院概况
学院概况

 师资队伍
师资队伍

 人才培养
人才培养

 招生就业
招生就业

 科学研究
科学研究

 平台基地
平台基地

 党群工作
党群工作

 校友之家
校友之家

 安全工作
安全工作

 网上办事
网上办事

 当前位置:
当前位置: